Functional genomics and DNA array techniques in atherosclerosis research
Journal 소개
이 논문은 1999년 Current Opinion in Lipidology에 게재된 논문으로 초창기 동맥경화(atherosclerosis)에 관한 연구를 DNA array와 접목, 소개함
Support by Kuopio University Hospital Uropean Union Biomed Program
DNA array
- 발병 기전에서 전사 단계에서 일어나는 기작들을 탐지하는 도구 (ex, atherosclerosis)
- 외가닥의 DNA가 nitrocellulose membrane에 강하게 결합, 상보적인 RNA와 hybridization
- 처음으로 고체 표면에 기초한 hybridization을 시도한 것은 DNA를 gel 상에서 분리한 Southern Blot임
- 작은 면적에 (micro) 수백 개의 oligonucleotide나 cDNA로 된 유전자 조각 (gene fragment)을 고밀도로 배열 (array)한 것을 의미. 즉, 유전자 검색을 위해 엄청나게 많은 종류의 DNA를 고밀도로 특정한 고형물 (주로 glass)에 붙여 놓은 것
- 응용 분야
1) 유전자 발굴(gene discovery), 암 분류(tumor classification), 유전자 발현 형태에 따른 질병 위험도 평가(risk assesment)
2) 질병 예후 진단(prognosis prediction), 질병진단(disease diagnosis), 신약 개발(drug discovery). 독성학연구(toxicological research)
3) 식품 안전성 평가, 유전병 연구 등
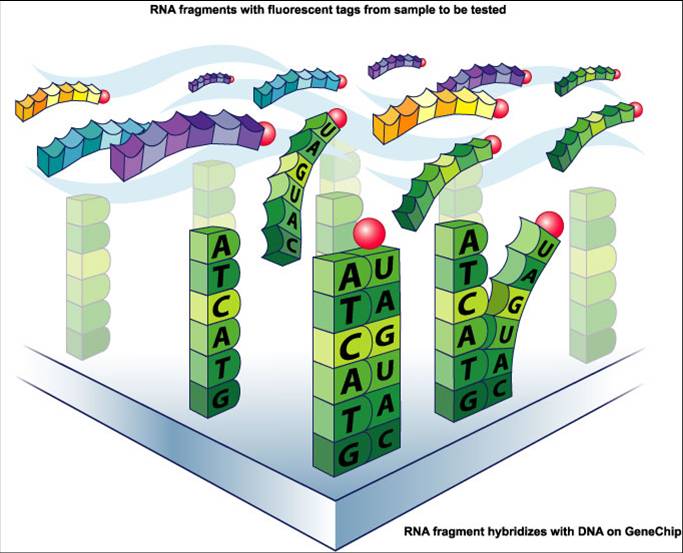
Fundamental basis of DNA microarray (complementary binding)
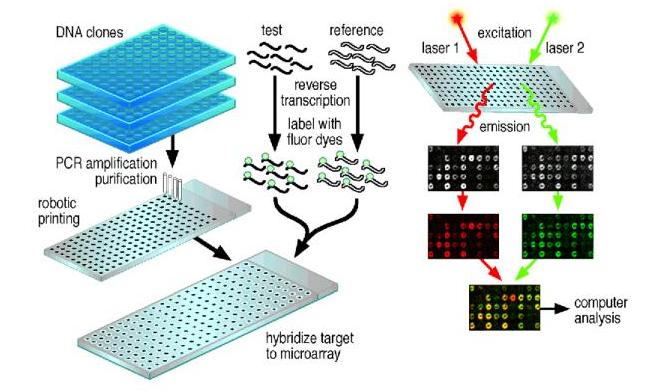
Southern Blots vs Northern Blots


DNA array = Massively parallel version of Southern & Northern blotting
Difference
- Southern, Northern Blot
; Distribute the oligonucleotide probes over a gel
Radioactively labeled oligonucleotide
"one gene, one messenger "
- DNA array
; Oligonucleotide probes are immobilized on a surface at micrometer distance
Sample is usually labeled with a fluorescent dye
" many genes, many messenger "
DNA microarray 종류 및 제작 방법
- 붙이는 유전 물질의 크기에 따라
1) Oligonucleotide microarray
- 보통 15~25개의 염기들로 이루어진 nucleotide가 microarray 구성
- 종류 : Affymetrix - photosynthesis를 통해 DNA microarray substrate, 즉 고형물 위에 직접 oligonucleotide 합성
Nanogen - oligonucleotide 합성한 후, 전기장을 이용해서 microarray 위의 특정 위치에 배열
2) cDNA microarray
- 최소한 500bp 이상의 유전자 조각이 붙여져 있음
- Stanford 대학에서 처음 개발됨, 주로 pin spotting과 inkjet 방식으로 만듬
논문에서 제시한 DNA array formats
Atherosclerosis
- A multifactorial disease of large, medium sized arteries
- Characterized by
smooth muscle cell migration
Proliferation
Accumulation of cholesterol
Formation of connective tissue
Presence of inflammatory cells
Thrombus formation
Calcification (석회성 물질의 침착)
- DNA array technology가 atherosclerosis를 비롯한 다양한 질병의 발병 기작을 연구하는 데에 혁신적인 역할을 할 수 있을 것이라고 주장함
Bioinformatics
- Transcriptome (즉, genome내에서의 모든 탐지 가능한 mRNA species의 level) 의 분석은 사용한 array 방법에 상관없이 연구자들에게 생물정보학적인 도구를 요구하기에 이르게 됨
- 엄청난 양의 data는 의미있는 방법을 이용하여 분석되고 표현되어져야 한다는 의견이 모아짐
- 이상적인 방법으로 유전자 발현 실험으로부터 얻은 데이터는 단일한 구조 (uniform structure)를 요구함으로써, 나온 결과들이 meta-analysis에 이용되도록 하는 것임
- 예를 들면 Genbank(http://www.ncbi.nlm.nih.gov) 처럼 유사한 구조를 가지는 centralized public data의 확립이 잠재적인 대안으로 나오게 됨
Application of DNA arrays in atherosclerosis research
- 이번 논문에서는 DNA array 기술을 이용하여 atherogenesis 동안 혈관벽의 유전자 발현 패턴을 분석
사용한 DNA array kit : Genomesystem’s (St Louis, MO) DNA filter arrays of 18000 cDNA clone
- 또한 산화된 LDL(low density lipoprotein)으로 자극을 준 후 내피 세포 반응 (endothelial cell response)을 transcriptional level에서 관찰
- 동맥에서의 sample은 심혈관 수술후, 또는 사체로부터 금방 얻음
- DNA array의 normalization은 모든 유전자의 발현 강도 (expression intensity)를 이용하여 수행함
Results

- 정상 혈관과 동맥경화 혈관의 차이점 -> 다른 세포 유형의 존재
- DNA array를 이용하여 cell culture에서의 유전자 발현 양상을 빠르게 screening할 수 있고, 또한 동정을 할 수 있게 됨
- 산화적으로 변형된 LDL입자들이 “내피세포의 기능장애를 유발하는 주요인자”
- DNA array 결과 왼쪽의 그림처럼 몇몇 유전자에서의 발현 정도(endothelial cell, atherosclerotic lesion)가 서로 다른 것을 알 수 있었음
Conclusion
- DNA array는 in vitro, in vivo 상에서의 DNA, RNA 발현을 조사할 수 있는 systemic한 도구라고 할 수 있음
- 가까운 미래에는 protein, antibody chip을 이용한 전사에서 단백질 발현으로의 생리학적, 병리학적 pathway를 밝힘으로써 DNA array data를 보충할 수 있는 data를 만들 수 있다고 봄